Développement d’un marqueur moléculaire pour l’identification des lamproies au stade juvénile
Il y a trois espèces de lamproie en France : la lamproie marine (Petromyzon marinus), la lamproie de rivière ou lamproie fluviatile (Lampetra fluviatilis) et la lamproie de Planer (L. planeri). La lamproie de Planer et la lamproie de rivière étant impossibles à distinguer morphologiquement au stade juvénile, les chercheurs ont développé une approche moléculaire qui permet aujourd’hui de discriminer ces deux espèces.
Introduction
La conservation de la lamproie marine (Petromyzon marinus), de la lamproie de rivière ou lamproie fluviatile (Lampetra fluviatilis) et de la lamproie de Planer (L.planeri) représentent un enjeu majeur. En effet, la lamproie marine est classée en tant qu’espèce en danger, et la lamproie de rivière est considérée comme vulnérable, selon les critères de l'Union internationale pour la conservation de la nature (UICN), sur la liste rouge des poissons de France. En outre, la répartition de ces trois espèces reste mal connue en France, et ce projet visait initialement à développer une approche de détection des trois espèces à partir d’ADN environnemental (ADNe).
À l’âge adulte, la lamproie marine peut-être distinguée facilement des deux autres espèces de lamproie (photo 1). Néanmoins, au stade juvénile, les larves dites « ammocètes » des trois espèces sont impossibles à distinguer dans leur première année. À partir d’une taille d’environ 5 cm, les ammocètes de P. marinus présentent une coloration sombre sur la nageoire caudale qui permet de les distinguer des larves des deux autres espèces. De plus, des marqueurs de l’ADN mitochondrial permettent aussi de distinguer P. marinus des deux espèces de Lampetra (Docker et al., 1999). En revanche, la lamproie fluviatile et la lamproie de Planer sont très proches génétiquement et leur ADN mitochondrial est similaire. De plus, il n’est pas possible de les distinguer morphologiquement à tous les stades juvéniles. À l’âge adulte, c’est principalement la taille qui permet de distinguer L. planeri (10 à 15 cm) de L. fluviatilis (18 à 30 cm). Ces deux espèces s’hybrident et sont considérées comme des écotypes
Photo 1. Photographie des trois espèces de lamproie présentes en France.

Nous avons tout d'abord identifié des marqueurs de type SNP (Single Nucleotide Polymorphism) avec des allèles distincts entre L. fluviatilis et L. planeri. En pratique, cela consiste à déterminer des zones de l’ADN avec des bases distinctes entre les deux espèces, parmi les quatre bases azotées possibles de l’ADN (adénine, thymine, guanine et cytosine). Nous avons utilisé des données avec plusieurs milliers de SNPs analysés sur 186 individus collectés en France lors d’une étude précédente (Rougemont et al., 2017). Cette analyse a nécessité l’assemblage d’un génome de lamproie de Planer. L’individu choisi a été un mâle collecté sur l’Oir, un cours d’eau de l’Observatoire de recherche en environnement sur les poissons diadromes dans les fleuves côtiers (ORE diaPFC). Une particularité des lamproies est que seules les cellules germinales possèdent la totalité du génome, l’ADN a donc été extrait à partir du sperme de cet individu. Nous avons ainsi détecté cinq SNPs discriminants entre L. fluviatilis et L. planeri. Parmi eux, nous en avons sélectionné un qui possède l’avantage de ne pas être présent chez P. marinus, sa non-amplification permettant de distinguer les genres Lampetra et Petromyzon.
Validation du marqueur
Nous avons ensuite développé un protocole de PCR
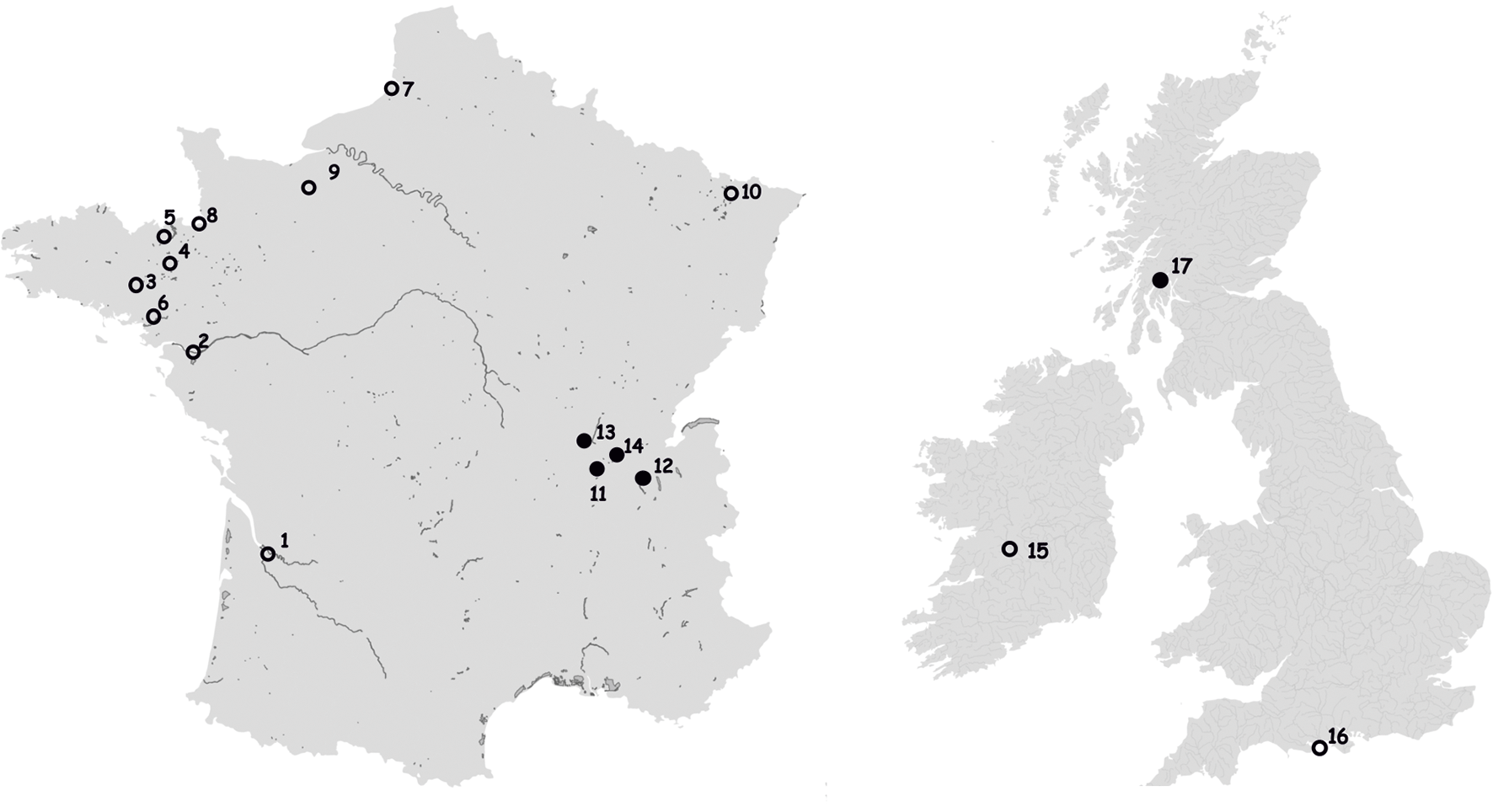
Les résultats ont été cohérents avec l'identification morphologique dans tous les cas, sauf pour les échantillons provenant du bassin du Rhône et du Loch Lomond en Écosse (Souissi et al., 2022). Dans le cas du Rhône, nous pensons que la présence d’une lignée très divergente de la lamproie de Planer (voire d’une espèce cryptique
Figure 1. Cartes des 17 sites échantillonnés.
Conclusion
En conclusion, à l’échelle nationale, le marqueur diagLpf permet de distinguer les larves ammocètes de L. planeri et L. fluviatilis (hors bassin du Rhône). Ainsi, lors de la capture d’ammocètes dans le cadre d’inventaires et de pêches électriques, l’identification de l’espèce est désormais possible à partir d’un morceau de nageoire. Cet outil permettrait donc de connaître la répartition des ammocètes des trois espèces de lamproies dans les bassins versants, et de déterminer une éventuelle différence d’habitats entres espèces en lien avec les caractéristiques physiques (profondeur, température, degré d’oxygénation, granulométrie…) et biotiques (présence d’autres espèces de lamproies ou d’invertébrés) du substrat. Nous avons également testé l’amplification de diagLpf avec des échantillons d’ADNe extraits à partir de prélèvements d’eau. Néanmoins, malgré de nombreuses tentatives et tests de divers protocoles, cette démarche n’a pas abouti. À l’avenir, les nombreux développements techniques en cours dans le domaine de l’ADNe, nous permettent toutefois d’espérer pouvoir détecter la présence des lamproies par cette approche.
Souissi, A., Besnard, A. L., Evanno, G. (2022). Développement d’outils moléculaires pour l’identification et l'étude de la distribution des trois espèces de lamproies à l'échelle nationale. Rapport final. OFB-Inrae- Institut Agro-UPPA. 17 p.
Photo d’entête : © Rostislav (Adobe Stock)
Notes
- 1. Les écotypes sont des groupes de populations d'une même espèce qui présentent des adaptations distinctes en réponse aux conditions environnementales spécifiques de leur habitat.
- 2. La Polymerase Chain Reaction ou PCR (ou encore ACP pour amplification en chaîne par polymérase), est une technique de réplication ciblée in vitro. Elle permet d'obtenir, à partir d'un échantillon complexe et peu abondant, d'importantes quantités d'un fragment d'ADN spécifique et de longueur définie.
- 3. En France, les associations migrateurs sont des associations « Loi 1901 » dont les missions sont la gestion, la protection et la restauration des stocks de poissons migrateurs.
- 4. Espèce difficile à distinguer morphologiquement ou écologiquement d'autres espèces apparentées, même si elle est génétiquement distincte.
- 5. Homozygote s'il possède deux copies identiques de l'allèle correspondant à un certain gène et hétérozygote si les deux allèles sont différents pour ce même gène.
Références
- Bernatchez, L., Guyomard, R., & Bonhomme, F. (1992). DNA sequence variation of the mitochondrial control region among geographically and morphologically remote European brown trout Salmo trutta populations. Molecular Ecology, 1(3), 161173. doi:10.1111/j.1365-294x.1992.tb00172.x
- Docker, M. F., Youson, J. H., Beamish, R. J., & Devlin, R. H. (1999). Phylogeny of the lamprey genus Lampetra inferred from mitochondrial cytochrome b and ND3 gene sequences. Canadian Journal of Fisheries and Aquatic Sciences, 56(12), 23402349. doi:10.1139/f99-171
- Evanno, G. (2017). Inferring the demographic history underlying parallel genomic divergence among pairs of parasitic and nonparasitic lamprey ecotypes. Molecular Ecology, 26(1), 142162. doi:10.1111/mec.13664
- Rougemont, Q., Gaigher, A., Lasne, E., Côte, J., Coke, M., Besnard, A. L., Launey, S., & Evanno, G. (2015). Low reproductive isolation and highly variable levels of gene flow reveal limited progress towards speciation between European river and brook lampreys. Journal of Evolutionary Biology, 28(12), 22482263. doi:10.1111/jeb.12750
- Rougemont, Q., Gagnaire, P. A., Perrier, C., Genthon, C., Besnard, A. L., Launey, S., & Evanno, G. (2017). Inferring the demographic history underlying parallel genomic divergence among pairs of parasitic and nonparasitic lamprey ecotypes. Molecular Ecology, 26(1), 142162. doi:10.1111/mec.13664
- Souissi, A., Besnard, A. L., & Evanno, G. (2022). A SNP marker to discriminate the European brook lamprey (Lampetra planeri), river lamprey (L. fluviatilis) and their hybrids. Molecular Biology Reports, 49(10), 1011510119. doi:10.1007/s11033-022-07800-8
Résumé
Il y a trois espèces de lamproie en France : la lamproie marine (Petromyzon marinus), la lamproie de rivière (Lampetra fluviatilis) et la lamproie de Planer (L.planeri). La lamproie marine est classée en danger et la lamproie de rivière vulnérable, selon les critères de l’Union internationale pour conservation de la nature (UICN), sur la liste rouge des poissons de France. La répartition des trois espèces reste mal connue en France, et ce projet visait initialement à développer une approche de détection des trois espèces à partir d’ADN environnemental (ADNe). Une première étape a été le développement de marqueurs moléculaires permettant de distinguer ces espèces. En particulier, la lamproie de Planer et la lamproie de rivière étant impossibles à distinguer morphologiquement au stade juvénile, ce projet a permis de développer un outil moléculaire de discrimination de ces deux espèces. Ainsi, il est désormais possible d’identifier l’espèce à partir d’un morceau de nageoire prélevé de manière non létale sur une larve (dite « ammocète ») collectée sur le terrain. Cette approche pourra permettre de mieux connaître la distribution de chaque espèce de lamproie et en particulier les zones de croissance des ammocètes qui passent plusieurs années enfouies dans le substrat meuble des cours d’eau.
Pas de document complémentaire pour cet article
Statistiques de l'article
 Vues: 5662
Vues: 5662
Téléchargements
 PDF: 161
PDF: 161
 XML: 34
XML: 34

